Des scientifiques indiens ont trouvé des insertions semblables au SIDA dans le coronavirus chinois
Des scientifiques indiens ont trouvé dans 2019nCov des insertions semblables au SIDA qui ne sont pas présentes dans les autres coronavirus. Cela laisse penser que le virus pourrait être le résultat de manipulations génétiques. L’étude a été publiée sur le site bioRxiv le 31 janvier 2020.
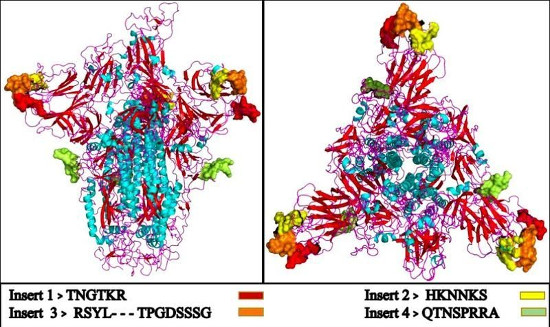
Glycoprotéine homo-trimère modélisée du virus 2019-nCoV. Les inserts de la protéine d’enveloppe du VIH sont représentés par des perles colorées, présentes au site de liaison de la protéine.
Traduction d’extraits du papier :
Résumé
Nous assistons à une épidémie majeure causée par le nouveau coronavirus 2019 (2019- nCoV). L’évolution du 2019-nCoV reste insaisissable. Nous avons trouvé 4 insertions dans la glycoprotéine de pointe (S) qui sont uniques au 2019-nCoV et ne sont pas présentes dans les autres coronavirus. Il est important de noter que les résidus d’acides aminés dans les 4 insertions ont une identité ou une similarité avec ceux du HIV-1 gp120 ou du HIV-1 Gag. Il est intéressant de noter que, bien que les inserts soient discontinus sur la séquence primaire d’acides aminés, la modélisation 3D du 2019-nCoV suggère qu’ils convergent pour constituer le site de liaison au récepteur.
La découverte de 4 inserts uniques dans le 2019-nCoV, qui ont tous une identité/similarité avec les résidus d’acides aminés dans les protéines structurelles clés du VIH-1 est peu susceptible d’être fortuite dans la nature.
Ce travail fournit des informations encore inconnues sur le 2019-nCoV et éclaire l’évolution et la pathogénicité de ce virus, avec des implications importantes pour le diagnostic de ce virus.
Analyse de l’évolution du 2019-nCoV
Il a été supposé que le 2019-nCoV est une variante du Coronavirus dérivée d’une source animale qui s’est transmise à l’homme. Compte tenu du changement de spécificité pour l’hôte, nous avons décidé d’étudier les séquences de la glycoprotéine de pointe (protéine S) du virus. Les protéines S sont des protéines de surface qui aident le virus à reconnaître l’hôte et à s’y fixer. Ainsi, une modification de ces protéines peut se traduire par un changement de la spécificité du virus pour l’hôte. Pour connaître les altérations du gène de la protéine S du 2019-nCoV et ses conséquences dans les réarrangements structurels, nous avons effectué une analyse in-sillico du 2019-nCoV par rapport à tous les autres virus. Un alignement multiple des séquences d’acides aminés de la protéine S du 2019-nCoV, de type Bat-SARS, SARS-GZ02 et MERS a révélé que la protéine S a évolué avec une diversité significative la plus proche de celle du SARS-GZ02 (Figure 1).
Figure 1
Insertions dans la région de la protéine Spike du 2019-nCoV
Comme la protéine S du 2019-nCoV partage l’ascendance la plus proche avec le SARS GZ02, les séquences codant pour les protéines spike de ces deux virus ont été comparées à l’aide du logiciel MultiAlin. Nous avons trouvé quatre nouvelles insertions dans la protéine de 2019-nCoV- « GTNGTKR » (IS1), « HKNNKS », « HKNKR » et « HKNKR ». (IS2), « GDSSSG » (IS3) et « QTNSPRRA » (IS4) (Figure 2). À notre grande surprise, ces insertions de séquences étaient non seulement absentes dans la protéine S du SRAS, mais n’ont été observées chez aucun autre membre de la famille des Coronaviridae (figure supplémentaire). Ceci est surprenant car il est très peu probable qu’un virus ait acquis naturellement de telles insertions uniques en peu de temps.

Figure 2
Les insertions présentent des similitudes avec le VIH
On a observé que les insertions étaient présentes dans toutes les séquences génomiques du virus 2019-nCoV disponibles dans les récents isolats cliniques. Pour connaître la source de ces insertions dans le 2019-nCoV, un alignement local a été effectué avec BLASTp en utilisant ces insertions comme interrogation avec tout le génome du virus. De manière inattendue, toutes les insertions ont été alignées avec le virus de l’immunodéficience humaine 1 (VIH 1).
Une analyse plus approfondie a révélé que les séquences alignées du VIH-1 avec le 2019-nCoV étaient dérivées de la glycoprotéine de surface gp120 (positions de la séquence d’acides aminés : 404-409, 462-467, 136-150) et de la protéine Gag (366-384 acides aminés). La protéine Gag du VIH est impliquée dans la liaison à la membrane de l’hôte, l’emballage du virus et la formation de particules semblables au virus. Gp120 joue un rôle crucial dans la reconnaissance de la cellule hôte en se liant au récepteur primaire CD4, ce qui induit des réarrangements structurels dans GP120, créant un site de liaison de haute affinité pour un co-récepteur de chimiokine comme CXCR4 et/ou CCR5.
yogaesoteric
31 mai 2020
